- Laboratorio
- Medicina di laboratorio
- Software di analisi
- SoftGenetics, LLC.
Software per sequenziamento NGS NextGENe®di analisida laboratorio
Aggiungi ai preferiti
Confronta con altri prodotti
Vuoi acquistare direttamente?
Vai sul nostro Shop.
Caratteristiche
- Funzione
- di analisi
- Applicazioni
- per sequenziamento NGS, da laboratorio
Descrizione
Il software NextGENe è il partner analitico perfetto per l'analisi dei dati di sequenziamento desktop prodotti dai sistemi Illumina® iSeq, Miniseq, MiSeq, NextSeq, HiSeq e NovaSeq, dai sistemi Ion Torrent Ion GeneStudio S5, PGM e Proton e da altre piattaforme.
Il software NextGENe viene eseguito su un sistema operativo Windows®, che offre un'interfaccia "point & click" di facile utilizzo per i biologi. Non richiede scripting o altri supporti bioinformatici, spesso necessari quando si utilizzano programmi come CLC Genomics Workbench, Lasergene SeqMan Pro e software accademici come MAQ & SOAP, Top Hat, BWA e Bowtie.
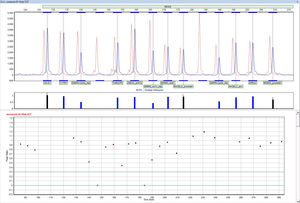
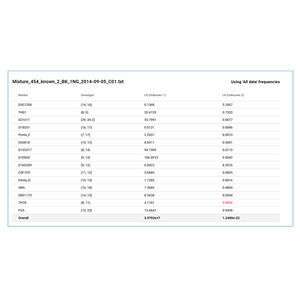
Il software NextGENe impiega tecnologie uniche e specifiche della piattaforma in un unico pacchetto multi-applicazione indipendente. Il software NextGENe contiene moduli di analisi per:
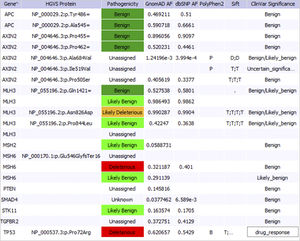
Rilevamento di SNP/Indel
Germinale
Somatico
Cattura dell'esoma, sequenziamento dell'intero esoma (WES)
Sequenziamento dell'intero genoma (WGS)
Analisi delle varianti strutturali (compreso il rilevamento dei geni di fusione)
Confronti tra famiglie e trio
Confronto tumore-normale
Rilevamento delle variazioni del numero di copie (CNV) e screening alternativo a quello MLPA
assemblaggio de novo
Trascrittoma; analisi dello splicing alternativo e livelli di espressione trascrizionale
ChIP-Seq, Espressione genica digitale (DGE), Analisi e quantificazione dei miRNA e Metagenomica
Il software NextGENe è stato progettato in un ambiente Windows® facile da usare per i biologi, riducendo in modo significativo la necessità di risorse bioinformatiche aggiuntive e i costi. Il software NextGENe utilizza un hardware a basso costo basato sul sistema operativo Windows a 64 bit. (Fare clic qui per la configurazione hardware suggerita)
---
Ricerche correlate
- Software medicale di analisi
- Software visualizzatore
- Software di controllo
- Software da laboratorio
- Software di monitoraggio
- Software automatizzato
- Software di progettazione
- Software con sistema di tracciatura
- Software d’importazione
- Software server
- Software da ricerca
- Software di interpretazione
- Software per biologia molecolare
- Software di genetica
- Software per sequenziamento NGS
- Software NGS
- Software per ricerca clinica
- Software per genomica
- Software per sequenziamento
- Software a elettroforesi capillare
* I prezzi non includono tasse, spese di consegna, dazi doganali, né eventuali costi d'installazione o di attivazione. I prezzi vengono proposti a titolo indicativo e possono subire modifiche in base al Paese, al prezzo stesso delle materie prime e al tasso di cambio.