Software di analisi GeneMarker®HTS di controlloper reportisticaNGS

Aggiungi ai preferiti
Confronta con altri prodotti
Vuoi acquistare direttamente?
Vai sul nostro Shop.
Caratteristiche
- Funzione
- di analisi, di controllo, per reportistica, NGS
- Applicazioni
- medico, per sequenziamento di DNA
Descrizione
Il software GeneMarker®HTS fornisce un flusso di lavoro semplificato e convalidato per i casi forensi di DNA mitocondriale, STR e Y-STR, nonché per la ricerca medica sul DNA mitocondriale da piattaforme di squenziamento parallelo massivo (MPS), come Illumina® e Ion Torrent®, in un sistema operativo Windows® di facile utilizzo.
Software di analisi forense GeneMarkerHTS (High Through-put Sequence) per dati provenienti da piattaforme di sequenze di nuova generazione (NGS) e sequenziamento massicciamente parallelo (MPS), tra cui Illumina® e Ion Torrent®
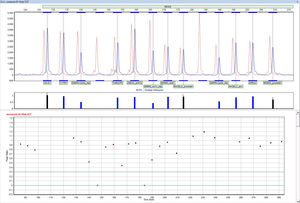
Analisi del DNA mitocondriale
Analisi dell'intero genoma, HVI/HVII e della regione di controllo
Tecnologia di allineamento unica ∙ Motif Consensus
Nomenclatura forense
Facile caricamento su EMPOP
Analisi STR
Autosomica e Y-STR
Nomenclatura forense
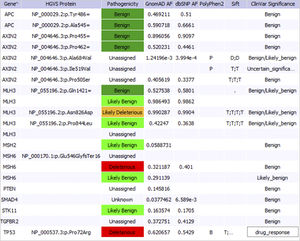
Segnalazione di genotipi e SNP
Concordante
Interfaccia Windows® convalidata e facile da usare
Compatibile con le principali chimiche e piattaforme
Traccia di controllo e controllo dell'utente
Opzioni di reportistica complete
Il software GeneMarkerHTS include:
Funzionalità di audit trail
Gestione degli utenti
Visualizzazione e reportistica personalizzabile per proteggere la privacy delle informazioni sanitarie personali (PHI)
Capacità di confronto dei profili
Caricamento su EMPOP
Analisi rapida: risultati in pochi minuti
mtDNA:
30 file di dati di cromosomi mtDNA interi MiSeq con profondità di copertura media di 10.000 sono stati allineati in 90 minuti (3 minuti per campione).
200 file di dati di cromosomi mtDNA interi con profondità di copertura media di 10.000 sono stati allineati in 16 ore.
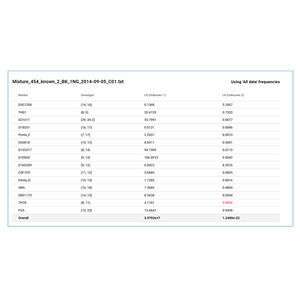
STR/Y-STR
I risultati del software GeneMarker HTS sono stati concordanti al 99,74% con le chiamate alleliche CE di 20.000 loci GeneMarker campionati.
Chiamate alleliche e iso-alleli dei campioni
---
VIDEO
Cataloghi
Nessun catalogo è disponibile per questo prodotto.
Vedi tutti i cataloghi di SoftGenetics, LLC.Ricerche correlate
- Software medicale di analisi
- Software di controllo
- Software da laboratorio
- Software per reportistica
- Software da ricerca
- Software per biologia molecolare
- Software di genetica
- Software NGS
- Software per ricerca clinica
- Software per genomica
- Software per sequenziamento
- Software a elettroforesi capillare
- Software per sequenziamento di DNA
* I prezzi non includono tasse, spese di consegna, dazi doganali, né eventuali costi d'installazione o di attivazione. I prezzi vengono proposti a titolo indicativo e possono subire modifiche in base al Paese, al prezzo stesso delle materie prime e al tasso di cambio.