- Laboratorio
- Biologia molecolare
- Modulo software di analisi
- JSI medical systems
Modulo software per sequenziamento NGS SEQNEXTdi analisidi acquisizionedi cartografia
Aggiungi ai preferiti
Confronta con altri prodotti
Vuoi acquistare direttamente?
Vai sul nostro Shop.
Caratteristiche
- Funzione
- di analisi, di acquisizione, di cartografia
- Applicazioni
- da laboratorio, per sequenziamento NGS
Descrizione
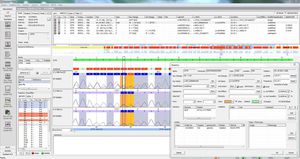
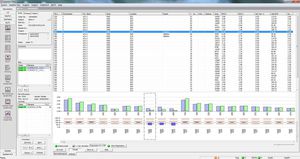
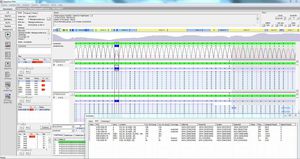
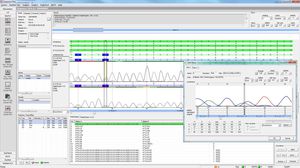
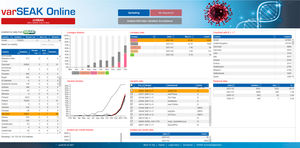
SEQNEXT è un'applicazione potente e di facile utilizzo per la mappatura, l'allineamento e l'individuazione delle varianti dei dati di sequenziamento di nuova generazione. SEQNEXT è in grado di analizzare i dati provenienti da tutte le piattaforme e i kit di sequenziamento più comuni (basati su PCR, arricchimento, cattura della sequenza). La visualizzazione di tutte le varianti rilevate, come delezioni, inserzioni e indel di qualsiasi lunghezza, SNP, regioni ripetute, CNV e fusioni geniche, è chiara e intuitiva.
SEQNEXT offre l'accesso a database pubblici di SNP come dbSNP, 1000 Genomes, ClinVar, ExAC e gnomAD per la classificazione e il filtraggio. Tutti i dati dei risultati possono essere scambiati con il nostro database delle varianti per la Shared Experience And Knowledge - varSEAK, trasferiti ai sistemi LIM interni del laboratorio e/o rilasciati come referti personalizzati per i pazienti.
NUOVA Analisi migliorata dei dati di sequenziamento dei virus, ad es. SARS-CoV-2
Trioanalisi e poolanalisi migliorata
Annotazioni dell'ontologia delle sequenze nella tabella delle varianti
Opzione di salto a varSEAK e a tutti i DB esterni disponibili, come dbSNP, ClinVar, UK10K, ESP, gnomAD ed ExAC
Analisi del sequenziamento dell'intero esoma (WES)
Compatibile con i dati di tutte le piattaforme e i kit di sequenziamento di nuova generazione più comuni
Facile impostazione di singole regioni target (ROI) tramite l'importazione di file bed o manifest basati su hg19 e/o hg38
Importazione di liste di geni per il filtraggio e l'analisi dei dati WES
Analisi di file fastq, fastq.gz o bam
Standard efficienti per la mappatura, l'allineamento, la qualità e la chiamata di varianti (algoritmi utilizzati: BWA e Smith-Waterman, adattati da JSI)
Impostazioni personalizzate per l'analisi e la chiamata di varianti (≥ 0,1%)
Elevata sensibilità e specificità per il rilevamento di delezioni, inserzioni e indel di qualsiasi lunghezza, SNP, regioni ripetute nonché CNV e fusioni geniche
---
Cataloghi
SEQUENCE PILOT
2 Pagine
Ricerche correlate
- Software medicale di analisi
- Software per reportistica
- Software automatizzato
- Modulo software
- Software d’importazione
- Software server
- Modulo software di analisi
- Software per biologia molecolare
- Modulo software da laboratorio
- Modulo software di acquisizione
- Modulo software di controllo
- Software NGS
- Modulo software di cartografia
* I prezzi non includono tasse, spese di consegna, dazi doganali, né eventuali costi d'installazione o di attivazione. I prezzi vengono proposti a titolo indicativo e possono subire modifiche in base al Paese, al prezzo stesso delle materie prime e al tasso di cambio.