- Laboratorio
- Medicina di laboratorio
- Software di gestione dati
- PhenoSystems SA

- Prodotti
- Cataloghi
- News & Trends
- Fiere
Software per sequenziamento NGS GensearchNGSdi analisidi gestione datidiagnostico
Aggiungi ai preferiti
Confronta con altri prodotti
Vuoi acquistare direttamente?
Vai sul nostro Shop.
Caratteristiche
- Funzione
- di analisi, di gestione dati, diagnostico
- Applicazioni
- per sequenziamento NGS, da laboratorio
Descrizione
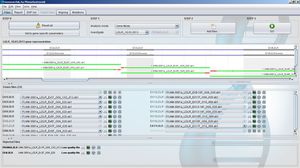
GensearchNGS è una soluzione software integrata per l'analisi di dati DNA-Seq provenienti da apparecchiature NGS di uso comune come Roche/454, Illumina, Ion Torrent e Proton. È stato progettato e sviluppato in stretta collaborazione con i principali gruppi diagnostici accreditati in Europa e con il supporto del progetto NMDchip FP7.
Integra tutti i passaggi dalla lettura della sequenza di importazione e di filtraggio, la suddivisione del codice a barre, l'allineamento, il rilevamento della variante e l'annotazione fino al rapporto finale delle varianti rilevate. Le varianti possono essere annotate con varie fonti di dati pubbliche e private (Ensembl, Alamut, Genome Trax, ...) Possono essere filtrate da combinazioni logiche tra campioni per studi familiari.
Caratteristiche principali:
Software di facile utilizzo per la ricerca di progetti NGS in un ambiente diagnostico, che gira su sistemi Windows, Linux e MacOS.
Si collega*) agli strumenti di interpretazione delle varianti e alle banche dati, come ad esempio: Ensembl, dbSNP, Alamut, Clinvar, Wiki Pathways, Gene Ontology*).
Filtrare le varianti tra i campioni (ad esempio varianti comuni a più campioni o escludere varianti di un campione da un altro).
Sistema di database centrico del paziente per la gestione dei dati di sequenza e delle varianti convalidate.
Possibilità di personalizzazione per l'esportazione dei dati nel vostro sistema di gestione del laboratorio.
Possibilità di pubblicare le vostre varianti su Clinvar*).
Sviluppato in collaborazione con laboratori accreditati.
Per progetti più grandi (WES/WGS): possibilità di utilizzare più PC in parallelo.
Tecnologia plug-in: allineamento con i nostri algoritmi proprietari o pubblici come Bowtie, BWA o Stampy.
Annotazione e filtraggio con descrittori e codici HPO, OMIM e Orphanet, utili per la ricerca di mutazioni in interi esomi o genomi interi.
---
* I prezzi non includono tasse, spese di consegna, dazi doganali, né eventuali costi d'installazione o di attivazione. I prezzi vengono proposti a titolo indicativo e possono subire modifiche in base al Paese, al prezzo stesso delle materie prime e al tasso di cambio.